「期待検証性能(expected validation performance)」をもとにしたパラメータ探索の検証
こんにちは@shunk031です。 温泉は素晴らしいですが、ホテルのインターネットが遅いと生きた心地がしないので人生難しいと感じています。
この記事は彌冨研 Advent Calendar 2019 19日目の記事です。

今回はAllenAiから公開されているハイパーパラメータ探索allentuneに実装されているアルゴリズムであるShow Your Work: Improved Reporting of Experimental Resultsを紹介します。
背景および導入
自然言語処理の研究において、しばしば提案手法が先行研究よりも優れたテストスコアを出すことによって提案手法の良さを実証するというロジックで論文が書かれます。この論文では、こうしたテストデータに対するスコアだけで手法の良し悪しを判断するのは難しいことを述べています。また、validationデータを用いたモデルの訓練で得られるスコアに焦点を当てて検証を行っています。
本論文では computation budget (例えばハイパーパラメータの探索回数やモデル全体の訓練時間) を関数として、ベストなモデルを発見するに至るまでの expected validation performance (期待検証性能) を示しています。こうした指標に従って筆者らが先行研究の再検証を行った結果、より多くの時間を掛けてモデルを訓練することで、報告されているスコアとは異なるスコアになる場合があることを表しています。
期待検証性能
異なるハイパーパラメータ を割り当てられた複数のモデル
の集合
を考えます。
あるハイパーパラメータ
で訓練されたモデルを
とします。
ハイパーパラメータを探索するにはたくさんの計算リソースが必要です。今回はそのリソースを表すような関数を computation budget
として定義しておきます。
最適なハイパーパラメータ が見つかったときに、そのパラメータを設定した
に対してテストデータにおけるスコアを論文に書きます。
ここで、
個のハイパーパラメータがある場合のベストなvalidationスコアは以下のように定義できます:
ここで、はtrainデータ
で学習させたモデル群
とそのハイパーパラメータ群
におけるvalidデータ
に対するvalidationスコアを返す関数です。
上記の式をもとに、異なるハイパーパラメータで何度も繰り返しvalidationスコアを計算したときに、の分布が得られます。
従って、期待性能
]として推定可能になります。
本論文では、実験結果を得るために必要な計算コストを概算する方法を導入します。
個のハイパーパラメータにおける期待性能は以下のように定義できます。
この指標を用いることで、あるスコアに到達するまでに必要な計算コストについて知ることができます。
特にvalidationスコアに対する期待性能を 期待検証性能 と呼んでいます。
ハイパーパラメータ探索を行う際の推奨事項
ハイパーパラメータの値を決める手法として、 grid search や uniform sampling が一般的に使われます。また適応的にハイパーパラメータをサンプリングするベイズ最適化も有名です。
こうしたハイパーパラメータ探索を検証するために、実験の再現性を担保するためのチェックリストが示されています。
特に本論文で導入されている 期待値検証性能 を基にしたパラメータ探索の検証を行うために必要な項目もあります。
再現性を担保するためのチェックリスト
研究者が報告する実験結果の再現性の向上のために、以下のようなチェックリストが論文内で提供されています。
- 実験結果の報告について
- 計算リソースについて
- 各アプローチの平均実行時間
- train/validationの分割の詳細情報
- 報告された各test結果に対応するvalidationのパフォーマンス
- 実装の公開リンク
- ハイパーパラメータサーチについて
- 各ハイパーパラメータの探索域
- ベストモデルにおけるハイパーパラメータのセッティング
- ハイパーパラメータ探索の試行回数
- ハイパーパラメータのサンプリング手法
- validationのパフォーマンスの期待値
EMNLP2018におけるチェックリストの集計結果
以下は上記のチェックリストに対する有無のパーセンテージを示しています。
- 実験結果の報告について
- 計算リソースについての有無: 18%
- 各アプローチの平均実行時間の有無: 14%
- train/validationの分割の詳細情報の有無: 92%
- 報告された各test結果に対応するvalidationのパフォーマンスの有無: 24%
- 実装の公開リンクの有無: 30%
- ハイパーパラメータサーチについて
- 各ハイパーパラメータの探索域の有無: 8%
- ベストモデルにおけるハイパーパラメータのセッティングの有無: 74%
- ハイパーパラメータ探索の試行回数の有無: 10%
- ハイパーパラメータのサンプリング手法の有無: 14%
- validationのパフォーマンスの期待値: 10%
特にハイパーパラメータの探索域や試行回数が論文中に記述されない場合が多いようです。最近は実装が公開されている論文も多く見られるように感じますが、それでも30%程度であることも驚きでした。
おわりに
今回、allentuneに実装されているShow Your Work: Improved Reporting of Experimental Resultsで提案されている 期待検証性能 について触れました。計算コストの観点から期待できる性能をもとに検証を行っていました。
ハイパーパラメータ探索の再現性を担保するために、計算リソースの規模やハイパーパラメータ探索関連の設定等のチェックリストが示されていました。 より大規模化するモデルとそのハイパーパラメータ探索をより再現しやすくするために、こうしたプロトコルで適切に評価し、再現できるような世界になるとよいですね。私自身もこうした点に注意したいと思います。
GitHub Actionsで最新のmecab-ipadic-neologd辞書を持つdocker imageをDocker Hubにpushする
こんにちは@shunk031です。草津温泉からこの記事を書いています。温泉は良いぞ。
この記事は彌冨研 Advent Calendar 2019 17日目の記事です。

私のレポジトリshunk031/mecab-neologd-py3では、最新のmecab-ipadic-neologdを持つpython3用のdocker imageをTravisCIを使って定期的にビルドし、Docker Hubにpushしています。 今回はGitHub Actions公開に伴い、TravisCIからの乗り換えを行います。
以下、2019年12月現在の情報です。
GitHub Actionsを有効にする
まずはGitHub Actionsを有効にします。 こちらから指示に従ってGitHub Actionsにアクセスできるようにします。
Sign up for the beta をクリックして進みます。

自身のユーザーアカウントが選択されている状態で Register for GitHub Actions Beta をクリックします。

はい、完了です。早いですね。では対象のレポジトリで Actions タブが現れていることを確認します。

Actions タブが追加されています。今後はこのタブから設定を追加していきます。

GitHub Actionsでdocker imageをビルドする
GitHub Actionsでdocker imageをビルドするワークフローを定義します。
Actions タブを選択すると、レポジトリの内容から自動的にマッチするワークフローがサジェストされています。今回は以下の Docker image ワークフローをベースに、docker imageをビルドするワークフローを定義します。

以下が完成したワークフローです。GitHub Actionsのワークフローはyaml形式で記述します。
name: Docker Image CI on: push: schedule: - cron: "0 0 * * 2,5" jobs: build: runs-on: ubuntu-latest steps: - uses: actions/checkout@master - name: Sign in Docker Hub run: docker login -u ${{ secrets.DOCKER_HUB_USERNAME }} -p ${{ secrets.DOCKER_HUB_PASSWORD }} - name: Build and push Docker image run: | docker build -t shunk031/mecab-neologd-py3 . export TAG=`date +%Y.%m.%d` docker tag shunk031/mecab-neologd-py3 shunk031/mecab-neologd-py3:$TAG docker push shunk031/mecab-neologd-py3:$TAG docker tag shunk031/mecab-neologd-py3 shunk031/mecab-neologd-py3:latest docker push shunk031/mecab-neologd-py3:latest
steps にワークフローの各ステップを記述します。今回Docker Hubにdocker imageをpushするにあたり、Docker Hubへのログイン と Docker imageのbuildとpush にステップを分けて実行させました。
Docker Hubへのログイン
Sign in Docker Hub と名前をつけたステップに、Docker Hubへのログイン処理を記述しました。ログインするにあたり、Docker Hubの username と password が必要になるため、Secrets 画面にて DOCKER_HUB_USERNAME と DOEKCER_HUB_PASSWORD を設定しました。これらの変数は表からは直接見えないように暗号化されます。

このようにSecretsで設定した変数はyamlファイル内で ${{ secrets.DOCKER_HUB_USERNAME }} のように参照することが可能です。
Docker imageのbuildとpush
Build and push Docker image と名前をつけたステップに、docker imageのビルド等の処理を記述しました。
私の場合、最新の辞書を持ったimageがlatestになるよう、またビルドした日付をタグに付与しています。日々新語や道語に対応し変化する辞書の任意の時点でのimageを取得したいというモチベーションです。
cronを用いた定期的なbuildとpush
mecab-ipadic-neologdは月曜と木曜に更新されます*1。したがって、その翌日の火曜と金曜に新しい辞書を取り込んだdocker imageのビルドを schedule の cron 機能で自動ビルドを実現します。
設定方法はlinuxのcronと同様のフォーマットで記述します。私はcronのフォーマットをいつもググって確認しているのですが、以下のようにポップアップで実行される時間が表示されるのでとても便利です。

(おまけ) READMEにバッジを付ける
ここまででGitHub Actionsで最新のmecab-ipadic-neologd辞書を持つdocker imageをDocker Hubにpushできるようにしました。おまけとしてワークフローが正しく実行されるかひと目で確認できるバッジをREADMEに追加します。

READMEにバッジを貼る際に必要なURLは https://github.com/(username)/(repository)/workflows/(workflow_name)/badge.svg で取得可能です。 workflow_name は ワークフローを記述するyaml の先頭にある name です。
READMEにバッジをつけるととてもそれっぽくなるので好きです。
おわりに
GitHub Actionsで最新のmecab-ipadic-neologd辞書を持つdocker imageをDocker Hubにpushするまでの工程を確認しました。GitHubがネイティブに提供するCI/CD環境はあたりまえですがGitHubと親和性が高く、これが無料*2で使えるというのは素晴らしいです。
Acknowledgement
mecab-ipadic-neologdにはいつもお世話になっております。作者の Toshinori Sato (@overlast)さま、ありがとうございます!
KDD'19 論文採択: マルチタスク学習とconditional attentionによる広告クリエイティブ評価
こんにちは@shunk031です。 Geforce RTX 2080Tiを50枚ほど使うとぽかぽかします。みなさんもぜひ試してみてください。
この記事は彌冨研 Advent Calendar 2019 2日目の記事です。

国際学会KDD2019に論文が採択されました。 KDDはデータマイニング分野で最も有名な会議です。 発表は8月にアラスカでありました。ポスター発表での採択です。 個人ブログにも記録として残そうと思います。
論文採択までの道のり
本論文はGunosyとの共同研究での成果となります。論文採択までの道のりは以下のGunosyデータ分析ブログに詳しく書きました。
KDD採択による外部発表
KDD採択に関連して、勉強会や学会に招待していただきました。
ICML/KDD 2019 Pre-conference session
LAPRASさん主催の ICML/KDD 2019 Pre-conference session にて、概要の発表を行いました。
LAPRAS @lapras_inc さんでICML/KDD採択者によるプレ発表会をやってきました!僕からもKDDで発表する内容を軽く説明させていただき、ポスターセッションでも議論させていただきました!素晴らしい会にお声がけくださったLAPRASの方々には感謝です。聞いてくださった皆様もありがとうございました😊 pic.twitter.com/UjXuW5mYjR
— しゅんけー (@shunk031) 2019年6月3日
テキストアナリティクスシンポジウム
第15回 テキストアナリティクスシンポジウムにて、依頼講演を行いました。 依頼講演のあとはリサーチインターン生とメンターを交えてパネルディスカッションの時間がありました。
参加レポート
KDDに参加した際の参加レポートを書きました。 僕は主に「広告xIT」や「解釈性・公正性」に関連したチュートリアルやワークショップに参加しました。
テンセントの広告技術が未来すぎる!AdKDD2019のテンセントAds招待講演まとめ
公平性および説明性を考慮した機械学習 in KDD2019
その他
KDD採択に関連して、いろんな方々に論文を読んでもらうことができました。 いくつかピックアップして以下に取り上げます。
arXivTimesさん
arXivTimesさんに取り上げてもらいました。ありがとうございます。
LINEさん
LINEさんの社内分析会議で取り上げてもらえました。ありがとうございます。
所感
今回採択された論文はたくさんの人の支えが合って執筆できたものです。締め切り前は必死に論文を書いていたのを今でも思い出します。 たくさんの思いがこもっているエモい論文がこうして素晴らしい学会に採択されて少しホッとしました。
沢山の人が支えてくれるので,僕はただ頑張るだけだと思って論文書いてます.たくさんの思いがこもってるエモい論文です.
— しゅんけー (@shunk031) 2019年1月28日
これからもたくさんの人とコラボレーションしながら、楽しく研究をやっていきます。
2018年の振り返り
こんにちは。@shunk031です。クリスマスにニューラルネットワーク"力"をお願いしましたが、何も貰えませんでした。サンタさんはHinton先生ではないみたいです。毎年同じお願いをしている気がします。
驚くべきことに2018年も今日で終わりみたいです。この1年何をやってきたか振り返りも兼ねて、思い出せるイベントについて軽く残しておこうと思います。
1月
2018年の目標をざっくり決めた
やりたいことをざっくり決めました。 shunk031.hatenablog.com
卒論執筆
卒論を書いてました。そんなに苦しまずに書けました。真面目に研究進めててよかったです。
言語処理学会予稿執筆
卒論でやっていたことを言語処理学会用に書き直していたりしました。こっちのほうが予稿が公に公開されるので先生のレビューが大変でした。初めてちゃんとした予稿を書いたので勉強になりました。
2月
頭痛が続く
2週間近く頭痛が続き、1週間ほどパソコンを触れないぐらいでした。病院で脳CTと血液検査を行いました。結果、脳は特に問題なかったのですが血液検査で肝臓が悪いことが判明して謎でした。処方された薬を飲んでいたら頭痛も治りました。人間の体って難しですね。
JSAI Cup 2018 人工知能学会データ解析コンペティションに参加
SIGNATEのクックパッドコンペに参加していました。去年よりもいい順位になったので少しは成長しているのでは説がありました。
www.slideshare.net
3月
言語処理学会 2018 in 岡山
岡山で開催された言語処理学会に参加しました。
10:30からの文書分類セッションB6-5で「CE-CLCNN: Character Encoderを用いたCharacter-level Convolutional Neural Networksによるテキスト分類」という発表をします!NLPに対してCVの手法を惜しげもなく使ったチャレンジングなモデルになってます.興味がある方はぜひ聞きに来てください! #NLP2018
— しゅんけー (@shunk031) 2018年3月14日
卒業旅行
言語処理学会終わりに岡山から広島、島根を回りました。
大学卒業
え、もう大学4年間終わるんですか?という感じでした。とても充実した4年間でした。
明日卒業式でスーツなのですが、いわゆる黒の就活カバンみたいなの持ってなくて黒のカバン探した結果です #OpenSUSE pic.twitter.com/Y9YSplmgS3
— しゅんけー (@shunk031) 2018年3月23日
FaberCompanyインターン
参加しました。3日で10万円もらえて最高でした。
第2回Cpaw AI Competition主催
主催しました。参加者の方々に満足していただけてよかったです。
4月
大学院入学
学部から引き続き法政大学の大学院に進学しました。同時期にGPUが40枚ぐらい一緒に入学してきました。
NVIDIA Deep Learning Seminnerに参加
参加しました。最先端の研究事例とかを聞けて楽しかったです。
5月
研究室運動会
毎年やってるんですが、めちゃくちゃ楽しいです。
AIPR2018執筆
卒論でやっていたことをまとめて国際学会に出そうということでやっていました。
Our #AIPR2018 workshop paper "End-to-End Text Classification via Image-based Embedding using Character-level Networks" is available on arXiv: https://t.co/05JmXldz0c
— しゅんけー (@shunk031) 2018年10月9日
We improved the performance of NLP using image-based data augmentation (used Random Erasing in this time).
6月
DLLabハッカソン参加
参加しました。当日にチームに振り分けられ、対象データセットを用いた識別器を作るハッカソンでした。Azure上でdockerを動かして識別器を構築する感じでした。途中dockerコンテナがハングして消し飛んでしまい、ボリュームもマウントしていなかったのもあってスナップショットも消えてしまいました。スコア的にはトップを取れそうだったので悲しかったです。このあとめちゃくちゃdocker勉強しました。
Piascore DLアドバイザー
DLLabハッカソンでSoTAを詰め込んだ手法でモデルを構築しているところを認められ、同じチームだったpiascoreさまにお話を頂いてdeep learning技術中心のアドバイザーを始めました。僕自信もとても成長できるディスカッションを複数回させていただき、とても勉強になりました。
7月
バイト
何社かサマーインターンを応募して受かっていたのですが、最終的には好きなことをやらせていただけて時給も他社と合わせていただけたgunosyで広告周りの研究開発をさせていただくことになりました。
ISIC2018コンペティション参戦
ISIC2018の皮膚障害自動診断システムのコンペティションに参加していました。このころ結構大変で辛かった思いがあります。そもそもメインでNLPをやっていたのでCV系の話で英語論文書くとは思っていなかったです。
8月
バイト
引き続き広告周りの研究開発をやっていました。そのままYANSに出そうという流れでエイヤでやりました。メンターさん、指導教員両方にとても迷惑を掛けてしまう感じになってしまったのですが結果をまとめられてよかったです。
HackU最優秀賞受賞
時間がない中進めていたHackUにて最優秀作品賞をいただくことができました。初めてみんなとチーム開発できたので楽しかったです。
YANS2018 in 香川
夏休みにgunosyでやっていたことをまとめてYANSで発表しました。うどんを食べまわったのをよく覚えています。
9月
Cpaw LT
LTで皮膚障害自動診断の最新動向について発表しました。
研究室合宿
今年は秩父でした。B4が卒論中間発表の練習をするのを応援するだけなので修士ポジションは楽でした。最終日雨の中ラフティングをやったのをよく覚えています。
10月
AIPR2018参加
初めての国際学会で、そもそも初めての海外でした。めちゃくちゃ楽しかったです。また論文書いて海外行きたいです。
第3回Cpaw AI Competition主催
主催しました。参加者の方々に満足していただけてよかったです。
11月
大学院の授業で論文読み
大学院のヒューマンインターフェースの授業で論文読みをやりました。
初めての国際学会も終わったところで、こんなことを考えてました。
ここ半年ずっと感じてる事なんですけど、少ないインプットに対してずっとアウトプットを吐き出し続けてる感じがしていて、実はもう何も吐き出せないのに無理してる感じがするんですよね…忙しいのはありがたい事だけど、もう少ししっかり勉強する時間を作らねばなぁと思いながら目の前のことに精一杯で
— しゅんけー (@shunk031) 2018年11月27日
12月
Kaggle Meetup Tokyo LT参加
これまで主催してきたCpaw AI Competitionの話をさせていただきました。各所から同様のことをやりたいというお話もあり、いろいろなフィードバックをいただくことができました。
アドベントカレンダー執筆
初めてアドベントカレンダーなるものに2つほど参加して記事を書きました。
修論中間発表
学会で発表したものをそのまま発表しました。難なく終わってよかったです。
国内学会論文執筆
情報処理学会2019と言語処理学会2019を並行して書いてます。大変なことになってます。
実装とか
Chainerを用いた再現実装をいくつか公開したりもしました。最近はPyTorchも書いたりしてます。
github.com github.com github.com github.com github.com github.com github.com github.com
まとめ
外部で発表することが多い1年でした。B4でインプットしたことを吐き続ける1年だったなとも感じます。来年はM2となり進路を考えなければならなくなったりと、純粋に研究だけに打ち込める時間が減りそうなのが心配です。引き続きインプット・アウトプットを大切にしていきます。
Xonshの設定を ~/.xonsh/*.py に分割して読み込む
こんにちは。shunk031です。この時期こたつに入って読む論文は最高ですよね。
この記事は Xonsh Advent Calendar 2018 17日目の記事です。
xonshの設定をする際、設定ファイルを複数に分割して読み込みたいなーと考えました。 そこでふと思ったのが「あれ?Pythonって先頭にドットがあるモジュールって読み込めるんだっけ?」という疑問です。
今回はホームディレクトリに .xonsh というドットから始まるディレクトリを作り、そこに分割した設定ファイル群をxonsh起動時に読み込む方法を調べました。
方法
まずは .xonshrc の設定例を示します。
import imp import os import sys XONSH_DIR = os.path.expanduser('~/.xonsh') sys.path.append(XONSH_DIR) open_file, file_name, desc = imp.find_module(XONSH_DIR) dot_xonsh = imp.load_module(XONSH_DIR, open_file, file_name, desc)
以上の方法で ~/.xonsh をモジュールとして読み込むことができます。
詳細
対象のディレクトリをPythonモジュールとしてインポートする
対象のディレクトリをPythonモジュールとしてインポートする場合は sys.path にディレクトリのパスを指定します。
import os import sys XONSH_DIR = os.path.expanduser('~/.xonsh') sys.path.append(XONSH_DIR)
先頭が ドット であるディレクトリを読み込む
ディレクトリ名の先頭が ドット から始まる場合、 imp モジュールを用いて以下のように読み込みを行います。
import imp
open_file, file_name, desc = imp.find_module(XONSH_DIR)
dot_xonsh = imp.load_module(XONSH_DIR, open_file, file_name, desc)
このようにして .xonsh ディレクトリを dot_xonsh モジュールとして読み込むことができます。
まとめ
Xonshの設定を ~/.xonsh/*.py に分割して読み込む際の「ドットから始まるモジュールの読み込み」の方法についてまとめました。imp モジュールを用いたモジュールのインポートを用いることでドットから始まるモジュールも読み込むことが可能となります。
ChainerCVを用いた皮膚障害検出システムの構築
こんにちは。shunk031です。NIPS改めNeurIPSが開催されてるということは12月ですね。この記事は Chainer/CuPy Advent Calendar 2018 12日目の記事です。

昨今はGoogleの本格的な医療分野への進出*1もあってか、医療xAIの分野に盛り上がりを感じます。 医療の現場では医師が目視で大量の診断画像を確認することが非常に多いです。 そこで今回は大量の診断画像から簡易的に病変部位を検出すべく、ChainerCVを用いた皮膚障害検出システムを構築していきたいと思います。
以降に皮膚障害画像等が含まれますので、苦手な方は 閲覧に注意して いただければ幸いです。
はじめに
近年Faster-RCNN*2やYOLO*3、SSD*4など、深層学習ベースの物体認識手法が高い認識精度を記録しています。今回はこうした物体認識手法のうち、SSD (Single Shot Multibox Detector) を用いて悪性度の高い皮膚障害であるメラノーマの領域を自動で検出するシステムをChainerCVを用いて実装しました。
なお、今回実装した結果は shunk031/chainer-skin-lesion-detector にて公開しております。
皮膚障害について
皮膚がんは皮膚障害の一種で、アメリカでは毎年5,000,000症例以上診断がなされています。特に悪性黒色腫「メラノーマ」*5は皮膚がんの最も深刻な形態で、皮膚がんにおける死亡率の多くを占めています。
2015年には、世界的なメラノーマの発症率は35万人を超えると推定され、約6万人が死亡したという報告があります。死亡率はかなり高いですが、早期発見の場合は生存率95%を超えています*6。したがって初期病徴を迅速に捉え、早期発見することが重要になってきます。
使用するデータセット
医療分野におけるデータ分析にする際に一番ネックとなるのがデータセットの少なさです。特に深層学習ベースの識別器を用いる際はとてもクリティカルな問題になります。今回のタスクである 皮膚障害検出 に対するデータセットも、「公開されていてそのまま使える」といったものは探した限りではありませんでした。
幸いながら、皮膚障害全般のデータセットは ISIC 2018 | ISIC 2018: Skin Lesion Analysis Towards Melanoma Detection という皮膚障害認識のコンペティションで用いられた、HAM10000*7*8という比較的規模の大きいデータセットがあります。
このHAM10000をベースに、今回のタスクである皮膚障害検出に対するデータセットを簡易的に作成することにしました。
以下の画像はHAM10000に含まれる、Lesion Segmentation (病変部位の輪郭検出) に用いられるデータの例です。

こうしたground truthに対して病変領域におけるバウンディングボックスを構築し、病変領域の検出を行おうと考えました。
皮膚障害検出データセットの作成
皮膚障害検出データセットを作成する際には以下2つのステップが必要です:
以下順を追って説明します。
バウンディングボックスの生成
セグメンテーション用のground truthから皮膚障害検出用のバウンディングボックスを取得します。PILのImage.getbbox*9を用いることで、画像内の非ゼロ領域のバウンディングボックスを計算できます。
In [1]: from PIL import Image In [2]: gt = Image.open('ISIC_0000000_segmentation.png') In [3]: gt.getbbox() Out[3]: (31, 29, 620, 438)
https://github.com/shunk031/chainer-skin-lesion-detector/blob/master/src/make_dataset.py#L72-L76
ChainerCVのutils*10とvisualizations*11を用いることで、簡単に画像の読み込みと可視化をして確認することができます。
ln [1]: from chainercv.utils import read_image ln [2]: from chainercv.visualizations import vis_bbox ln [3]: import numpy as np ln [4]: left, upper, right, lower = gt.getbbox() ln [5]: gt_img = read_image('ISIC_0000000_segmentation.png') ln [6]: vis_bbox(gt_img, np.asarray([[upper, left, lower, right]]))
実行すると以下のようにバウンディングボックスを含めて可視化することができます。

Image.getbboxを使うことで綺麗にバウンディングボックスを生成することができました。
次に、得られたバウンディングボックスをもとにVOCフォーマットのXMLファイルを作成します。
VOCフォーマットのXMLファイルの生成
先行研究の深層学習ベースの物体検出・認識アルゴリズムはPascal VOC*12といった大規模なデータセットを元に学習を行っています。 今回はこのPascal VOCのアノテーションデータと同じフォーマットのXMLファイルを作成し、それらをもとに学習を行っていきます。 ChainerCVはPascal VOCデータセットを想定した便利メソッドが多数揃っているため、恩恵も受けやすいです。
以下のブログを参考に、VOCフォーマットのXMLファイルを生成してみます。
In [1]: import xml.etree.ElementTree as ET In [2]: def make_voc_based_xml(folder_name, file_name, bbox): ...: """ ...: Make VOC based XML string ...: """ ...: left, upper, right, lower = bbox ...: annotation = ET.Element('annotation') ...: ...: annotation = ET.Element('annotation') ...: tree = ET.ElementTree(element=annotation) ...: folder = ET.SubElement(annotation, 'folder') ...: filename = ET.SubElement(annotation, 'filename') ...: objects = ET.SubElement(annotation, 'object') ...: name = ET.SubElement(objects, 'name') ...: pose = ET.SubElement(objects, 'pose') ...: truncated = ET.SubElement(objects, 'truncated') ...: difficult = ET.SubElement(objects, 'difficult') ...: bndbox = ET.SubElement(objects, 'bndbox') ...: xmin = ET.SubElement(bndbox, 'xmin') ...: ymin = ET.SubElement(bndbox, 'ymin') ...: xmax = ET.SubElement(bndbox, 'xmax') ...: ymax = ET.SubElement(bndbox, 'ymax') ...: ...: folder.text = folder_name ...: filename.text = file_name ...: name.text = 'lesion' ...: pose.text = 'frontal' ...: truncated.text = '1' ...: difficult.text = '0' ...: xmin.text = str(left) ...: ymin.text = str(upper) ...: xmax.text = str(right) ...: ymax.text = str(lower) ...: ...: return annotation
https://github.com/shunk031/chainer-skin-lesion-detector/blob/master/src/make_dataset.py#L20-L53
実行すると以下のようなXMLファイルを生成することができます。
<?xml version="1.0" ?> <annotation> <folder>ISIC2018_Task1_Training_GroundTruth</folder> <filename>ISIC_0000000_segmentation.png</filename> <object> <name>lesion</name> <pose>frontal</pose> <truncated>1</truncated> <difficult>0</difficult> <bndbox> <xmin>31</xmin> <ymin>29</ymin> <xmax>620</xmax> <ymax>438</ymax> </bndbox> </object> </annotation>
こうして得られたアノテーションデータと皮膚障害画像から、皮膚障害の検出を行うモデルのトレーニングを行います。
モデルのトレーニング
今回はChainerCVのSSDを用いて、以上で作成したアノテーションデータを元にモデルのトレーニングを行います。SSDのアルゴリズムの説明は今回省かせていただきます。実装に先立ちまして、ChainerCVをベースとした以下の資料がとても参考になりました。
ChainerCVを用いたSSDモデルの構築
ChainerCVのexampleを元にSSDを学習させるコードを記述します。具体的な実装は以下を御覧ください。
https://github.com/shunk031/chainer-skin-lesion-detector/blob/master/src/main.py
Chainer・ChainerCVは素晴らしい深層学習フレームワークなので特に躓くところはありません。もともとのexampleが素晴らしすぎるため、大部分をそのまま使い回すことができます。少し補足するならば、画像を読み込んで前処理をし、モデルに流すDatasetMixinクラスです。
import xml.etree.ElementTree as ET import chainer import numpy as np from chainercv.utils import read_image from util import const class ISIC2018Task1Dataset(chainer.dataset.DatasetMixin): def __init__(self, img_fpaths, gt_fpaths): assert len(img_fpaths) == len(gt_fpaths), \ f'# of image: {len(img_fpaths)} != # of ground truth: {len(gt_fpaths)}' self.annotations = self.load_annotations(img_fpaths, gt_fpaths) def load_annotations(self, img_fpaths, gt_fpaths): annotations = [] for img_fpath, gt_fpath in zip(img_fpaths, gt_fpaths): anno_dict = self.parse_annotation(gt_fpath) annotations.append((img_fpath, anno_dict)) return annotations def parse_annotation(self, xml_fpath): anno_dict = {'bbox': [], 'label': []} anno_xml = ET.parse(str(xml_fpath)) for obj in anno_xml.findall('object'): bndbox = obj.find('bndbox') name = obj.find('name').text.strip() anno_dict['label'].append(const.LABELS.index(name)) anno_dict['bbox'].append([ int(bndbox.find(tag).text) - 1 for tag in ('ymin', 'xmin', 'ymax', 'xmax')]) return anno_dict def __len__(self): return len(self.annotations) def get_example(self, i): img_fpath, anno_dict = self.annotations[i] img = read_image(str(img_fpath), color=True) bbox = np.asarray(anno_dict['bbox'], dtype=np.float32) label = np.asarray(anno_dict['label'], dtype=np.float32) # Return arrays with the following shape # img: (ch, h, w) # bbox: (num bboxes, (ymin, xmin, ymax, xmax)) # label: (num labels, ) return img, bbox, label
chainer.datasets.DatasetMixin クラスを継承し、get_exampleにて画像とバウンディングボックス、対象ラベルを返すようにする必要があります。
モデルの評価
予測精度
学習して得られたモデルを元に評価を行います。物体検出で広く用いられている評価指標である mean Average Precision (mAP) を元に予測精度を評価します。
from chainercv.evaluations import eval_detection_voc test_dataset = ISIC2018Task1Dataset(test, test_gt) # テストデータの準備 chainer.serializers.load_npz('model_best.npz', model) # 予め学習したモデルの読み込み result = {'ap': [], 'map': []} for idx in tqdm(range(len(test_dataset))): img, gt_bboxes, gt_labels = test_dataset[idx] # テストデータからデータを取得 bboxes, labels, scores = model.predict([img]) # モデルに予測させる score = eval_detection_voc(bboxes, labels, scores, [gt_bboxes], [gt_labels]) # mAP等を計算 result['ap'].append(score['ap']) result['map'].append(score['map']) print(np.mean(result['map'])) >> 0.9421965317919075
以上を実行した結果、今回構築したモデルにおいて mAPは 0.94 を記録しました。
予測結果の可視化
構築した皮膚障害検出システムは比較的高い精度を記録することができました。では実際のモデルの予測結果を見てみましょう。
成功例
以下に正しく検出できた例を示します。撮影の際に写り込んでしまう黒い枠に反応することなく、正しく病変部位を予測することができています。写り込んでしまっている体毛にも頑健であることが分かります。
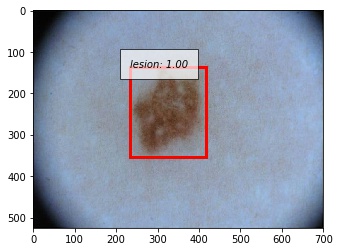
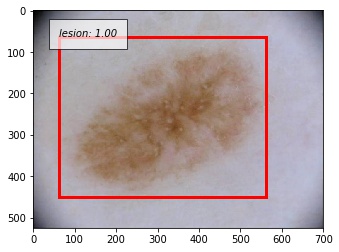
失敗例
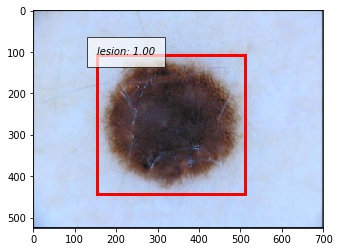
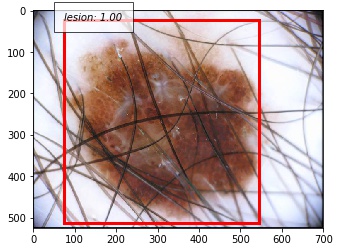
では次に正しく検出できなかった例を示します。システムがエラーしているサンプルを観察するのはとても重要です。
こちらは予測したバウンディングボックスがground truthよりも大きかった例です。
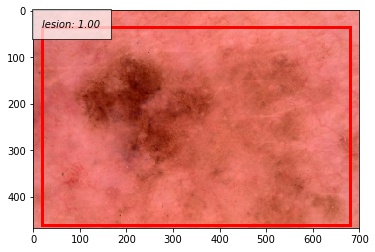

こちらは病変部位の色が薄いパターンです。
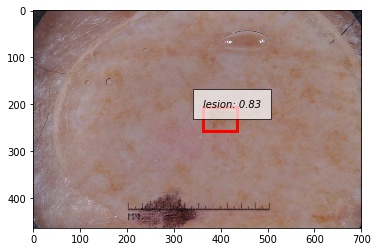
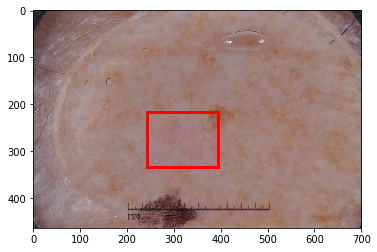
人間が診断しても正確な判断が困難なサンプルであることが見て取れると思います。こうしたサンプルに対してどのようにモデルを改善していくかが今後の課題になってきそうです。
おわりに
ChainerCVを用いて皮膚障害検出システムを構築しました。医療画像分野は取得できるデータがとても少なく、工夫をしてデータセットを作る場面があるかもしれません。そこで今回は他のタスクに利用可能な形でデータセットを簡易的に作成し、最新のモデルで病変部位の識別を行うシステムを構築しました。ChainerCVはとても簡単にモデルを作成することが可能で、なおかつ分析する際にもユーティリティーがとても便利に使えます。 医療xAIの分野は盛り上がりつつあります。PyTorchベースの医療診断フレームワーク等*13*14も現れてきておりますし、これからの発展がとても楽しみです。
参考
- 2018年現在の、最新の皮膚障害分類に関する資料です。
- 医療画像診断 研究動向2018 “ISIC 2018 Challenge”に見る皮膚障害認識 / A survey of medical image diagnosis in ISIC 2018 Skin Lesion Analysis Towards Melanoma Detection https://speakerdeck.com/shunk031/a-survey-of-medical-image-diagnosis-in-isic-2018-skin-lesion-analysis-towards-melanoma-detection
- 医療画像全般とてもまとまっている資料です。
- 日本メディカルAI学会公認資格:メディカルAIコースのオンライン講義資料です。深層学習を医療分野にどう適用するかの実践的な内容も含まれています。
- メディカルAIコース オンライン講義資料 —ドキュメント https://japan-medical-ai.github.io/medical-ai-course-materials/
*1:Scalable and accurate deep learning with electronic health records | npj Digital Medicine https://www.nature.com/articles/s41746-018-0029-1
*2:Ren, Shaoqing, et al. "Faster r-cnn: Towards real-time object detection with region proposal networks." Advances in neural information processing systems. 2015.
*3:Redmon, Joseph, et al. "You only look once: Unified, real-time object detection." Proceedings of the IEEE conference on computer vision and pattern recognition. 2016.
*4:Liu, Wei, et al. "Ssd: Single shot multibox detector." European conference on computer vision. Springer, Cham, 2016.
*5:5. 悪性黒色腫(メラノーマ)|一般社団法人日本皮膚悪性腫瘍学会 http://www.skincancer.jp/citizens_skincancer05.html
*6:About melanoma: https://challenge2018.isic-archive.com/
*7:Noel C. F. Codella, David Gutman, M. Emre Celebi, Brian Helba, Michael A. Marchetti, Stephen W. Dusza, Aadi Kalloo, Konstantinos Liopyris, Nabin Mishra, Harald Kittler, Allan Halpern: “Skin Lesion Analysis Toward Melanoma Detection: A Challenge at the 2017 International Symposium on Biomedical Imaging (ISBI), Hosted by the International Skin Imaging Collaboration (ISIC)”, 2017
*8:Tschandl, P., Rosendahl, C. & Kittler, H. The HAM10000 dataset, a large collection of multi-source dermatoscopic images of common pigmented skin lesions. Sci. Data 5, 180161 doi:10.1038/sdata.2018.161
*9:Image Module — Pillow (PIL Fork) 4.1.1 documentation https://pillow.readthedocs.io/en/4.1.x/reference/Image.html#PIL.Image.Image.getbbox
*10:Utils — ChainerCV 0.11.0 documentation https://chainercv.readthedocs.io/en/stable/reference/utils.html
*11:Visualizations — ChainerCV 0.11.0 documentation https://chainercv.readthedocs.io/en/stable/reference/visualizations.html
*12:The PASCAL Visual Object Classes Homepage http://host.robots.ox.ac.uk/pascal/VOC/
*13:perone/medicaltorch: A medical imaging framework for Pytorch https://github.com/perone/medicaltorch
*14:pfjaeger/medicaldetectiontoolkit: The Medical Detection Toolkit contains 2D + 3D implementations of prevalent object detectors such as Mask R-CNN, Retina Net, Retina U-Net, as well as a training and inference framework focused on dealing with medical images. https://github.com/pfjaeger/medicaldetectiontoolkit
ワシントンDCで行われた国際会議で発表してきて、そのノリでニューヨークとボストンに行ってきた 4/4
こんにちは。@shunk031です。段々と寒くなってきましたね。足元にGPUマシンだけが暖かく見守ってくれる季節になりました。
初めての国際学会でワシントンDCに3泊4日、その後移動してニューヨークに3泊4日、ボストンに3泊4日ほど観光してきました。 そもそも海外渡航が初めてだったので、準備等も含めて旅行記のようなものを残しておきます。

目次
学会発表
http://shunk031.hatenablog.com/entry/aipr2018_01shunk031.hatenablog.com
観光
なんとか初めての国際学会での発表を終えて、これからが本番です。ワシントンDC、ニューヨーク、ボストンの主要観光スポットをいい感じにまわる戦略でやっていきました。 観光するにあたり、Google mapのマイマップ機能はとても重宝しました。 dekiru.net
学会中は研究費で宿代などが出ていましたが、観光時にはAirbnbを使って格安で部屋をとりました。 abnb.me
移動には基本地下鉄を使いました。Pasmo/Suicaのような地下鉄カードを買っていい感じにチャージするとスムーズに乗れました。 夜まで観光してしまった場合はUberを使いました。初めて使ったんですがとても便利ですね。
https://www.uber.com/invite/epgfsxwww.uber.com
初日のワシントンダレス国際空港からホテルへは乗り合いのシャトルバスSuper Shuttleを使ってみました。 行き先を予め予約しておくとかなり格安で市街地のホテルまで乗せて行ってくれます。 私は予約せずにのりばのお姉さんに行き先を言って乗車したのですが、それでもUberの半額程度で目的地まで乗せてくれました。
ワシントンDC
学会発表終わりのワシントンDC観光編はこちら。
http://shunk031.hatenablog.com/entry/aipr2018_02shunk031.hatenablog.com
ニューヨーク
ニューヨーク観光編はこちら。とても刺激的な街でした。
http://shunk031.hatenablog.com/entry/aipr2018_03shunk031.hatenablog.com
ボストン
泊まったところ
- Central Square駅から5分ぐらいのところでとてもアクセスが良かったです。すぐ近くに綺麗なコインランドリーがあり、洗濯もできました。
食べたもの
Union Oyster House


Lobster Scampi, Mussels
 Live Lobsters Prepared Boiled Or Broiled
Live Lobsters Prepared Boiled Or Broiled
 Union Grilled Oysters
Union Grilled Oysters
らーめん山頭火

なるほど〜 pic.twitter.com/k8sJDwYYAw
— しゅんけー (@shunk031) October 16, 2018
Headhall
 Fish & Frites
Fish & Frites
 Buffalo Fried Shrimp
Buffalo Fried Shrimp
 New England Clam Chowder
New England Clam Chowder
- MITの近くのお店です。とても賑わっていました。ビールの種類がとても多いみたいです。
Boston Chowda Co.
Lobster Roll & Lobster Pieロブスターロールとロブスターパイ、ありえん極上ロブスターを楽しめる / I'm at Boston Chowda Company - @bostonchowdaco in Boston, MA pic.twitter.com/M1VbsEElTB
— しゅんけー (@shunk031) October 17, 2018
- ロブスターロールが美味しいお店と聞いてランチできました。サクッと食べれていいのですが、$25ぐらいでびっくりしました。
Boston and Maine Fish Co.
- ロブスターロールだけだと物足りなかったのでシュリンプカクテルを購入しました。
Summer Shack

 LOBSTERS IN THE ROUGH
LOBSTERS IN THE ROUGH


LOBSTER POTSTICKERS & MUSSELS
行ったところ
- Quincy Market
- Harvard University
- Massachusetts Institute of Technology
- The Boston Public Library
- Prudential Center Tower
ハーバード大学と日本のオタク pic.twitter.com/x5EKgeZhqp
— しゅんけー (@shunk031) 2018年10月16日
こちら、MITです pic.twitter.com/X0tUa4uArB
— しゅんけー (@shunk031) 2018年10月16日