ChainerCVを用いた皮膚障害検出システムの構築
こんにちは。shunk031です。NIPS改めNeurIPSが開催されてるということは12月ですね。この記事は Chainer/CuPy Advent Calendar 2018 12日目の記事です。

昨今はGoogleの本格的な医療分野への進出*1もあってか、医療xAIの分野に盛り上がりを感じます。 医療の現場では医師が目視で大量の診断画像を確認することが非常に多いです。 そこで今回は大量の診断画像から簡易的に病変部位を検出すべく、ChainerCVを用いた皮膚障害検出システムを構築していきたいと思います。
以降に皮膚障害画像等が含まれますので、苦手な方は 閲覧に注意して いただければ幸いです。
はじめに
近年Faster-RCNN*2やYOLO*3、SSD*4など、深層学習ベースの物体認識手法が高い認識精度を記録しています。今回はこうした物体認識手法のうち、SSD (Single Shot Multibox Detector) を用いて悪性度の高い皮膚障害であるメラノーマの領域を自動で検出するシステムをChainerCVを用いて実装しました。
なお、今回実装した結果は shunk031/chainer-skin-lesion-detector にて公開しております。
皮膚障害について
皮膚がんは皮膚障害の一種で、アメリカでは毎年5,000,000症例以上診断がなされています。特に悪性黒色腫「メラノーマ」*5は皮膚がんの最も深刻な形態で、皮膚がんにおける死亡率の多くを占めています。
2015年には、世界的なメラノーマの発症率は35万人を超えると推定され、約6万人が死亡したという報告があります。死亡率はかなり高いですが、早期発見の場合は生存率95%を超えています*6。したがって初期病徴を迅速に捉え、早期発見することが重要になってきます。
使用するデータセット
医療分野におけるデータ分析にする際に一番ネックとなるのがデータセットの少なさです。特に深層学習ベースの識別器を用いる際はとてもクリティカルな問題になります。今回のタスクである 皮膚障害検出 に対するデータセットも、「公開されていてそのまま使える」といったものは探した限りではありませんでした。
幸いながら、皮膚障害全般のデータセットは ISIC 2018 | ISIC 2018: Skin Lesion Analysis Towards Melanoma Detection という皮膚障害認識のコンペティションで用いられた、HAM10000*7*8という比較的規模の大きいデータセットがあります。
このHAM10000をベースに、今回のタスクである皮膚障害検出に対するデータセットを簡易的に作成することにしました。
以下の画像はHAM10000に含まれる、Lesion Segmentation (病変部位の輪郭検出) に用いられるデータの例です。

こうしたground truthに対して病変領域におけるバウンディングボックスを構築し、病変領域の検出を行おうと考えました。
皮膚障害検出データセットの作成
皮膚障害検出データセットを作成する際には以下2つのステップが必要です:
以下順を追って説明します。
バウンディングボックスの生成
セグメンテーション用のground truthから皮膚障害検出用のバウンディングボックスを取得します。PILのImage.getbbox*9を用いることで、画像内の非ゼロ領域のバウンディングボックスを計算できます。
In [1]: from PIL import Image In [2]: gt = Image.open('ISIC_0000000_segmentation.png') In [3]: gt.getbbox() Out[3]: (31, 29, 620, 438)
https://github.com/shunk031/chainer-skin-lesion-detector/blob/master/src/make_dataset.py#L72-L76
ChainerCVのutils*10とvisualizations*11を用いることで、簡単に画像の読み込みと可視化をして確認することができます。
ln [1]: from chainercv.utils import read_image ln [2]: from chainercv.visualizations import vis_bbox ln [3]: import numpy as np ln [4]: left, upper, right, lower = gt.getbbox() ln [5]: gt_img = read_image('ISIC_0000000_segmentation.png') ln [6]: vis_bbox(gt_img, np.asarray([[upper, left, lower, right]]))
実行すると以下のようにバウンディングボックスを含めて可視化することができます。

Image.getbboxを使うことで綺麗にバウンディングボックスを生成することができました。
次に、得られたバウンディングボックスをもとにVOCフォーマットのXMLファイルを作成します。
VOCフォーマットのXMLファイルの生成
先行研究の深層学習ベースの物体検出・認識アルゴリズムはPascal VOC*12といった大規模なデータセットを元に学習を行っています。 今回はこのPascal VOCのアノテーションデータと同じフォーマットのXMLファイルを作成し、それらをもとに学習を行っていきます。 ChainerCVはPascal VOCデータセットを想定した便利メソッドが多数揃っているため、恩恵も受けやすいです。
以下のブログを参考に、VOCフォーマットのXMLファイルを生成してみます。
In [1]: import xml.etree.ElementTree as ET In [2]: def make_voc_based_xml(folder_name, file_name, bbox): ...: """ ...: Make VOC based XML string ...: """ ...: left, upper, right, lower = bbox ...: annotation = ET.Element('annotation') ...: ...: annotation = ET.Element('annotation') ...: tree = ET.ElementTree(element=annotation) ...: folder = ET.SubElement(annotation, 'folder') ...: filename = ET.SubElement(annotation, 'filename') ...: objects = ET.SubElement(annotation, 'object') ...: name = ET.SubElement(objects, 'name') ...: pose = ET.SubElement(objects, 'pose') ...: truncated = ET.SubElement(objects, 'truncated') ...: difficult = ET.SubElement(objects, 'difficult') ...: bndbox = ET.SubElement(objects, 'bndbox') ...: xmin = ET.SubElement(bndbox, 'xmin') ...: ymin = ET.SubElement(bndbox, 'ymin') ...: xmax = ET.SubElement(bndbox, 'xmax') ...: ymax = ET.SubElement(bndbox, 'ymax') ...: ...: folder.text = folder_name ...: filename.text = file_name ...: name.text = 'lesion' ...: pose.text = 'frontal' ...: truncated.text = '1' ...: difficult.text = '0' ...: xmin.text = str(left) ...: ymin.text = str(upper) ...: xmax.text = str(right) ...: ymax.text = str(lower) ...: ...: return annotation
https://github.com/shunk031/chainer-skin-lesion-detector/blob/master/src/make_dataset.py#L20-L53
実行すると以下のようなXMLファイルを生成することができます。
<?xml version="1.0" ?> <annotation> <folder>ISIC2018_Task1_Training_GroundTruth</folder> <filename>ISIC_0000000_segmentation.png</filename> <object> <name>lesion</name> <pose>frontal</pose> <truncated>1</truncated> <difficult>0</difficult> <bndbox> <xmin>31</xmin> <ymin>29</ymin> <xmax>620</xmax> <ymax>438</ymax> </bndbox> </object> </annotation>
こうして得られたアノテーションデータと皮膚障害画像から、皮膚障害の検出を行うモデルのトレーニングを行います。
モデルのトレーニング
今回はChainerCVのSSDを用いて、以上で作成したアノテーションデータを元にモデルのトレーニングを行います。SSDのアルゴリズムの説明は今回省かせていただきます。実装に先立ちまして、ChainerCVをベースとした以下の資料がとても参考になりました。
ChainerCVを用いたSSDモデルの構築
ChainerCVのexampleを元にSSDを学習させるコードを記述します。具体的な実装は以下を御覧ください。
https://github.com/shunk031/chainer-skin-lesion-detector/blob/master/src/main.py
Chainer・ChainerCVは素晴らしい深層学習フレームワークなので特に躓くところはありません。もともとのexampleが素晴らしすぎるため、大部分をそのまま使い回すことができます。少し補足するならば、画像を読み込んで前処理をし、モデルに流すDatasetMixinクラスです。
import xml.etree.ElementTree as ET import chainer import numpy as np from chainercv.utils import read_image from util import const class ISIC2018Task1Dataset(chainer.dataset.DatasetMixin): def __init__(self, img_fpaths, gt_fpaths): assert len(img_fpaths) == len(gt_fpaths), \ f'# of image: {len(img_fpaths)} != # of ground truth: {len(gt_fpaths)}' self.annotations = self.load_annotations(img_fpaths, gt_fpaths) def load_annotations(self, img_fpaths, gt_fpaths): annotations = [] for img_fpath, gt_fpath in zip(img_fpaths, gt_fpaths): anno_dict = self.parse_annotation(gt_fpath) annotations.append((img_fpath, anno_dict)) return annotations def parse_annotation(self, xml_fpath): anno_dict = {'bbox': [], 'label': []} anno_xml = ET.parse(str(xml_fpath)) for obj in anno_xml.findall('object'): bndbox = obj.find('bndbox') name = obj.find('name').text.strip() anno_dict['label'].append(const.LABELS.index(name)) anno_dict['bbox'].append([ int(bndbox.find(tag).text) - 1 for tag in ('ymin', 'xmin', 'ymax', 'xmax')]) return anno_dict def __len__(self): return len(self.annotations) def get_example(self, i): img_fpath, anno_dict = self.annotations[i] img = read_image(str(img_fpath), color=True) bbox = np.asarray(anno_dict['bbox'], dtype=np.float32) label = np.asarray(anno_dict['label'], dtype=np.float32) # Return arrays with the following shape # img: (ch, h, w) # bbox: (num bboxes, (ymin, xmin, ymax, xmax)) # label: (num labels, ) return img, bbox, label
chainer.datasets.DatasetMixin クラスを継承し、get_exampleにて画像とバウンディングボックス、対象ラベルを返すようにする必要があります。
モデルの評価
予測精度
学習して得られたモデルを元に評価を行います。物体検出で広く用いられている評価指標である mean Average Precision (mAP) を元に予測精度を評価します。
from chainercv.evaluations import eval_detection_voc test_dataset = ISIC2018Task1Dataset(test, test_gt) # テストデータの準備 chainer.serializers.load_npz('model_best.npz', model) # 予め学習したモデルの読み込み result = {'ap': [], 'map': []} for idx in tqdm(range(len(test_dataset))): img, gt_bboxes, gt_labels = test_dataset[idx] # テストデータからデータを取得 bboxes, labels, scores = model.predict([img]) # モデルに予測させる score = eval_detection_voc(bboxes, labels, scores, [gt_bboxes], [gt_labels]) # mAP等を計算 result['ap'].append(score['ap']) result['map'].append(score['map']) print(np.mean(result['map'])) >> 0.9421965317919075
以上を実行した結果、今回構築したモデルにおいて mAPは 0.94 を記録しました。
予測結果の可視化
構築した皮膚障害検出システムは比較的高い精度を記録することができました。では実際のモデルの予測結果を見てみましょう。
成功例
以下に正しく検出できた例を示します。撮影の際に写り込んでしまう黒い枠に反応することなく、正しく病変部位を予測することができています。写り込んでしまっている体毛にも頑健であることが分かります。
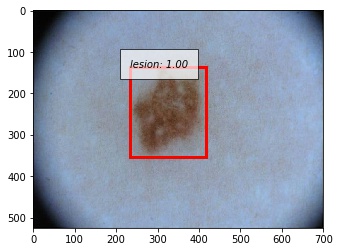
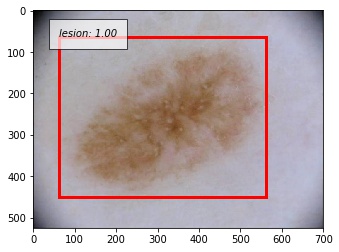
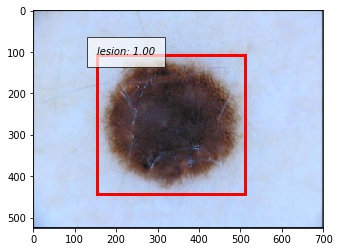
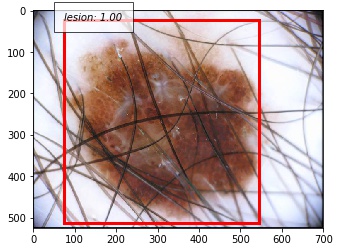
失敗例
では次に正しく検出できなかった例を示します。システムがエラーしているサンプルを観察するのはとても重要です。
こちらは予測したバウンディングボックスがground truthよりも大きかった例です。
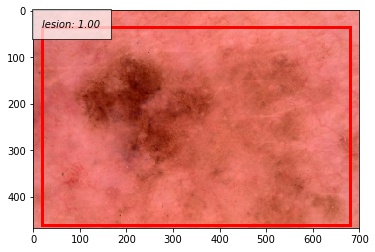

こちらは病変部位の色が薄いパターンです。
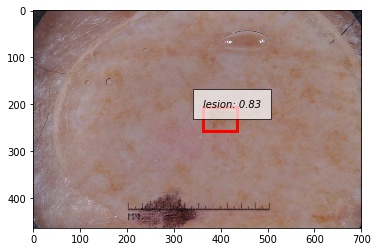
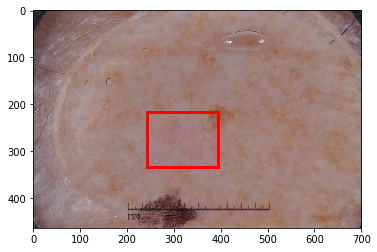
人間が診断しても正確な判断が困難なサンプルであることが見て取れると思います。こうしたサンプルに対してどのようにモデルを改善していくかが今後の課題になってきそうです。
おわりに
ChainerCVを用いて皮膚障害検出システムを構築しました。医療画像分野は取得できるデータがとても少なく、工夫をしてデータセットを作る場面があるかもしれません。そこで今回は他のタスクに利用可能な形でデータセットを簡易的に作成し、最新のモデルで病変部位の識別を行うシステムを構築しました。ChainerCVはとても簡単にモデルを作成することが可能で、なおかつ分析する際にもユーティリティーがとても便利に使えます。 医療xAIの分野は盛り上がりつつあります。PyTorchベースの医療診断フレームワーク等*13*14も現れてきておりますし、これからの発展がとても楽しみです。
参考
- 2018年現在の、最新の皮膚障害分類に関する資料です。
- 医療画像診断 研究動向2018 “ISIC 2018 Challenge”に見る皮膚障害認識 / A survey of medical image diagnosis in ISIC 2018 Skin Lesion Analysis Towards Melanoma Detection https://speakerdeck.com/shunk031/a-survey-of-medical-image-diagnosis-in-isic-2018-skin-lesion-analysis-towards-melanoma-detection
- 医療画像全般とてもまとまっている資料です。
- 日本メディカルAI学会公認資格:メディカルAIコースのオンライン講義資料です。深層学習を医療分野にどう適用するかの実践的な内容も含まれています。
- メディカルAIコース オンライン講義資料 —ドキュメント https://japan-medical-ai.github.io/medical-ai-course-materials/
*1:Scalable and accurate deep learning with electronic health records | npj Digital Medicine https://www.nature.com/articles/s41746-018-0029-1
*2:Ren, Shaoqing, et al. "Faster r-cnn: Towards real-time object detection with region proposal networks." Advances in neural information processing systems. 2015.
*3:Redmon, Joseph, et al. "You only look once: Unified, real-time object detection." Proceedings of the IEEE conference on computer vision and pattern recognition. 2016.
*4:Liu, Wei, et al. "Ssd: Single shot multibox detector." European conference on computer vision. Springer, Cham, 2016.
*5:5. 悪性黒色腫(メラノーマ)|一般社団法人日本皮膚悪性腫瘍学会 http://www.skincancer.jp/citizens_skincancer05.html
*6:About melanoma: https://challenge2018.isic-archive.com/
*7:Noel C. F. Codella, David Gutman, M. Emre Celebi, Brian Helba, Michael A. Marchetti, Stephen W. Dusza, Aadi Kalloo, Konstantinos Liopyris, Nabin Mishra, Harald Kittler, Allan Halpern: “Skin Lesion Analysis Toward Melanoma Detection: A Challenge at the 2017 International Symposium on Biomedical Imaging (ISBI), Hosted by the International Skin Imaging Collaboration (ISIC)”, 2017
*8:Tschandl, P., Rosendahl, C. & Kittler, H. The HAM10000 dataset, a large collection of multi-source dermatoscopic images of common pigmented skin lesions. Sci. Data 5, 180161 doi:10.1038/sdata.2018.161
*9:Image Module — Pillow (PIL Fork) 4.1.1 documentation https://pillow.readthedocs.io/en/4.1.x/reference/Image.html#PIL.Image.Image.getbbox
*10:Utils — ChainerCV 0.11.0 documentation https://chainercv.readthedocs.io/en/stable/reference/utils.html
*11:Visualizations — ChainerCV 0.11.0 documentation https://chainercv.readthedocs.io/en/stable/reference/visualizations.html
*12:The PASCAL Visual Object Classes Homepage http://host.robots.ox.ac.uk/pascal/VOC/
*13:perone/medicaltorch: A medical imaging framework for Pytorch https://github.com/perone/medicaltorch
*14:pfjaeger/medicaldetectiontoolkit: The Medical Detection Toolkit contains 2D + 3D implementations of prevalent object detectors such as Mask R-CNN, Retina Net, Retina U-Net, as well as a training and inference framework focused on dealing with medical images. https://github.com/pfjaeger/medicaldetectiontoolkit